تکنیک توالی یابی 454 | تکنولوژی 454 | پایروسکوئنسینگ
تکنولوژی Roche/454 Life Science
شرکت Roche/454 Life Science اولین موسسه پیشگام در تولید و تجاری سازی تکنولوژی NGS می باشد. این شرکت، در اواخر دهه ۱۹۹۰ برای اولین بار از تکنولوژی موسوم به پایروسکوئنسینگ (Pyrosequencing) برای تعیین توالی DNA در مقیاس وسیع استفاده کرد. در این سیستم، نمونه ی DNA به قطعاتی با طول حدود ۱۰۰ جفت باز شکسته می شود. سپس، دو آداپتور کوتاه به انتهای قطعات متصل می شود. این آداپتورها، محل اتصال پرایمرهای لازم برای تکثیر و توالی یابی هستند.
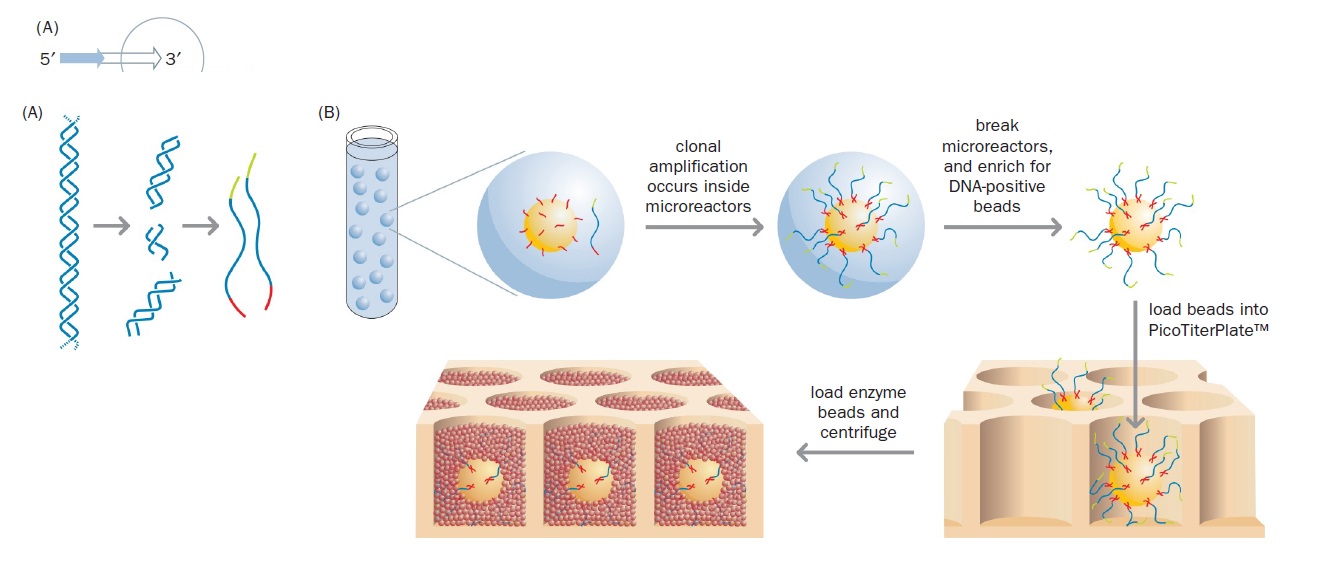
این قطعات، با ذرات کوچکی که پوششی از جنس استرپتاویدین در سطح خود دارند، مخلوط می شوند و به کمک آداپتور B خود که در انتهای ۵ بیوتینه است، به این ذرات متصل می گردند. آن گاه، قطعات دو رشته ای متصل به ذرات، دناتوره و تک رشته ای می گردند. این مخلوط، به اندازه ی کافی رقیق است؛ به طوری که به هر ذره ی منفرد، تنها یک رشته ی DNA متصل می باشد. سپس، با افزودن ترکیبات لازم برای تکثیر قطعات DNA متصل به ذرات، این مخلوط به امولسیون تبدیل می شود. به این ترتیب، هر ذره با رشته ی DNA متصل به آن، در یک قطره ی امولسیون به دام می افتد و واکنش Polymerase chain reaction (PCR) به طور مستقل در هر قطره انجام می گردد (Emulsion PCR).
در مرحله ی بعد، به منظور تعیین توالی، ذرات حاوی DNA بر روی صفحه ی سلیکونی متشکل از ۴۰۰۰۰۰۰ چاهک منظم با حجمی در حد پیکولیتر پخش می شوند (۱۱). اندازه ی کوچک چاهک، باعث می شود تا در هر چاهک بیش از یک ذره قرار نگیرد (شکل 1). سپس آنزیم های لازم یعنی DNA پلیمراز، سولفوریلاز و لوسیفراز به چاهک افزوده می شوند. چاهک ها به طور مجزا پی در پی در معرض dGIP ، dTTP ،dCTP، ATP قرار می گیرند و بین هر بار افزودن سوبسترای دئوکسی نوکلئوتید، یک مرحله ی شستشو انجام می شود. بدیهی است که ورود هر نوکلئوتید به رشته ی در حال گسترش، وابسته به حضور باز مکمل در الگو می باشد. با ورود هر نوکلئوتید مکمل، یک مولکول پیرو فسفات رها می شود. آنزیم سولفوریلاز، این مولکول پیرو فسفات را با استفاده از آدنوزین فسفوسولفات (Adenosine phosphosulfate یا APS) به ATP تبدیل می کند. آنزیم لوسیفراز نیز به کمک ATP حاصل شده، سوبسترای خود را که لوسیفیرین می باشد به اکسی لوسیفرین تبدیل می کند. نور ساطع شده توسط دوربین های متصل به صفحه سیلیکونی ثبت و به صورت پیک در یک گراف نمایان می شود (شکل 1).
رونقی و همکاران، در سال ۱۹۹۸ از پایه گذاران پایروسکوئنسینگ بودند (۱۲). در آخرین نسخه ی ارایه شده از طرف شرکت Roch، این تکنولوژی می تواند تا ۱۴ گیگاباز (Giga bases ; Gb ) توالی با طول متوسط ۷۰۰ جفت باز تولید کند. طول بلند توالیهای تولید شده توسط این روش و سرعت بالا، از مهم ترین ویژگیهای این تکنولوژی میباشد. هر اجرای پایروسکوئنسینگ بسیار سریع است؛ در واقع، این تکنیک می تواند تعداد زیادی اجرا را به طور همزمان انجام دهد. به عنوان مثال، ۹۶ اجرای مختلف می تواند به طور همزمان در یک پلیت ۹۶ چاهکی انجام شود، از آن جایی که کل فرایند اتوماتیک است، نیاز چندانی به توجه فرد آزمایش کننده ندارد. بدیهی است هر تکنولوژی در کنار مزایای خود، یک سری معایب نیز دارد و یکی از معایب این روش، درصد خطای به نسبت بالای آن در نواحی هموپلیمر DNA است. هزینه ی بالای توالی یابی با این روش در مقایسه با سایر روش های توالی یابی در مقیاس وسیع، عیب دیگر این تکنولوژی به شمار می رود که استفاده از آن را تا حدی محدود می کند (۱۳).
تکنولوژی ۴۵۴، توالی های بلندتری را در مقایسه با روش های دیگر توالی یابی در مقیاس وسیع تولید می کند. این مسأله، دقت و سر هم نمودن این توالی ها را به توالی های بلندتر را به طور قابل توجهی افزایش می دهد. با این وجود، چون تعداد خوانش های تولید شده در
هر اجرای این دستگاه اندک است، استفاده از آن برای موجوداتی با اطلاعات ژنتیک محدود، مستلزم اجرای متعدد می باشد که از نظر اقتصادی چندان مقرون به صرفه نیست. در مقابل، این تکنیک ابزاری قدرتمند برای توالی یابی مجدد ژنوم های شناخته شده می باشد (۱۱). به عنوان مثال، این روش برای توالی یابی بخش هایی از ژنهای یک شخص، به منظور تشخیص جهش های بیماری زا به خوبی عمل می کند. به منظور افزایش سرعت در این مورد، نوکلئوتیدها می توانند به همان ترتیبی که در توالی طبیعی شناخته شده وجود دارند، اضافه شوند. به این ترتیب، جهش از طریق عدم توانایی نوکلئوتید طبیعی برای اتصال در یک موقعیت خاص به سادگی تشخیص داده می شود (۱۰).
تکنیک توالی یابی ۴۵۴
قسمت دوم
Massively parallel pyrosequencing در سال ۲۰۰۵ توسط شرکت ۴۵۴ Life Sciences توسعه یافت و توسط Roche تجاریسازی شد. این تکنیک، توالی یابی ۴۵۴ نام دارد و نخستین تکنیک از توالی یابیهای نسل جدید است. توالی یابی ۴۵۴ میتواند بیش از یک میلیارد باز DNA را در یک روز تعیین توالی کند (معادل یک سوم ژنوم انسان). اولین گام در این تکنولوژی، آمادهسازی DNA به منظور تکثیر توسط PCR است. در واقع این تکنیکها به جای این که از محصولات PCR یا توالیهای کلونشده استفاده کنند، کار خود را با DNA ژنومی آغاز مینمایند. DNA ژنومی طبق پروتکل استاندارد استخراج DNA، از ارگانیسم موردنظر استخراج و سپس DNA خالص با استفاده از هموژنایزر اولتراسونیک (طی فرایند sonication) (و یا با استفاده از آنزیمهای محدودکننده)، به قطعات کوچکتری با اندازه ۳۰۰ تا ۵۰۰ جفتباز تقسیم میشود.
مقالات مرتبط:
NGS | توالی یابی نسل جدید | next generation sequencing
تکنولوژی ABI / SOLD
به منظور تکثیر هر کدام از قطعات، انتهایشان باید دارای توالی شناختهشدهای باشد و این موضوع خصوصا در ژنومهایی که هرگز توالی یابی نشدهاند، غیرممکن است. حتی اگر توالی شناخته شده باشد نیز سونیکاسیون به صورت تصادفی DNA را میشکند و راهی برای شناخت دقیق توالی هر کدام از انتهاها وجود ندارد. راه حلی که برای دور زدن این مشکل به کار گرفته میشود، استفاده از linker ها یا آداپتورها است که قطعات DNA کوتاهی با توالی شناخته شده میباشند. اتصال دو آداپتورمتفاوت به انتهاهای قطعات DNA صورت میگیرد و پس از دناتوراسیون، مولکولهای تکرشتهای دارای آداپتورهای متفاوت در دو انتها، گزینش میشوند. آداپتورها دو وظیفه بر عهده دارند: نخست اینکه باعث اتصال قطعات به bead ها میشوند و دوم اینکه به عنوان مکانی برای اتصال پرایمر عمل میکنند. درنتیجه پرایمر یکسانی برای تمام قطعات میتواند استفاده شود.
مرحله بعدی، اتصال مولکولهای انتخاب شده به bead ها است و با استفاده از مکانیسم استرپتاویدین-بیوتین انجام میپذیرد. پروتئین باکتریایی استرپتاویدین تمایل اتصال بالایی به ویتامین بیوتین دارد و با طراحی یکی از دو آداپتور به نحوی که در انتهای ۵’ خود دارای تگ بیوتین باشد، مولکولهای DNA به bead های پوشیده شده با استرپتاویدین متصل خواهند شد. به این نحو مولکولهای الگوی تکرشتهای DNA روی beadها ثابت میشوند و بعدا قطعات تولیدشده طی PCR نیز به سطح آن متصل خواهند شد.
جداسازی bead ها از یکدیگر با تشکیل امولسیون روغن در آب انجام میگیرد، طوری که هر قطره حاوی یک bead و واکنشدهندههای لازم برای PCR (دئوکسی نوکلئوتیدهای آزاد، پرایمرهای مکمل آداپتور و Taq پلیمراز) باشد. این موضوع در این تکنیک بسیار حیاتی است. این قطرهها microreactor نام دارند. وجود امولسیون، مانع از پراکنده شدن مولکولهای DNA و واکنشدهندهها از یک bead به سایر beadها میگردد.
پس از اتمام PCR، حدود ده میلیون نسخه از هر قطعه DNA به صورت ثابتشده بر روی هر bead وجود خواهد داشت. در مرحله بعدی امولسیون از هم پاشیده میشود و beadها در چاههای picoliter که روی یک اسلاید قرار دارند، رسوب داده میشوند، به نحوی که هر چاه حاوی یک bead باشد. سطح تحتانی چاهها شفاف است و نور تولید شده میتواند از آن عبور کرده و توسط دستگاه شناسایی شود.
چاهها سپس با bead های کوتاهتری که سطحشان دارای ATP سولفوریلاز و لوسیفراز است، پوشانده میشوند. پیشسازهای dNTP یکبهیک و با ترتیب مشخصی (T، سپس A، سپس C، سپس G) به bead ها افزوده میشوند و همزمان با تکثیر، خوانش توالیها صورت میگیرد (sequencing by synthesis)؛ به این نحو که با هر بار اتصال نوکلئوتید درست، نور نشرشده در تمام واکنشها شناسایی و ضبط میگردد. شدت نور تولیدشده منطبق بر تعداد نوکلئوتیدهایی از یک نوع است که به رشته الگو متصل شدهاند. به عنوان مثال شدت نوری که در اثر اتصال سه باز A ایجاد میشود، سه برابر حالتی است که یک باز A وجود دارد.
تکنیک Massively parallel sequencing شرکت ۴۵۴ Life Sciences، در هر واکنش توالی یابی، توالیهای نسبتا بلندی را میخواند و به علاوه هزاران عدد از چنین واکنشهایی را به طور موازی انجام میدهد. در نتیجه محصول نهایی حدود ۱۰هزار بار بزرگتر از توالی یابی دی دئوکسی میگردد. البته ایراداتی نیز در این تکنیک وجود دارد؛ از جمله اینکه تشخیص تعداد بازها در حالتی که چندین باز تکراری به دنبال هم میآیند (مثلا AAAAAA) دشوار است.


با سلام
در حالت توالی یابی کل اگزوم، چجوری اگزون ها رو از کل ژنوم جدا میکنیم؟ یعنی دو مسیر برای توالی یابی نسل اینده تعیرف شده که یکیش مسیر توالی یابی کل ژنومی هست و کل دی ان ای در اون مورد استفاده قرار میگیره ولی برای مسیر بعدی که توالی یابی اگزوم هست چجوری ما فقط اگزون ها رو استفاده میکنیم؟ ایا از طریق انزیم ریورس ترنسکریپتاز انجام میشه؟ و تولید cdna? ممنون میشم توضیح بدید👍
سلام وقت بخیر
سوال داشتم درمورد اینکه در مرحله آخر که همه چی انجام شده حالا چطور ترکیب توالی ها فهمیده میشه؟ یعنی اینکه چطور میشه فهمید قطعه ها چطور پشت سرهم قرار میگیرن؟
ممنون میشم پاسخ بدید🙏🏻
قاعدتا اگر اشتباه نکنگ هر کدوم از توالی ها مناطق همپوشان با توالی قبل و بعد خود خواهند داشت که در مرحله آخر کمک می کنه ترتیب توالی ها را تشخیص بدهیم