
کلیات طراحی پروتئین یا مهندسی پروتئین :
طراحی پروتئین یا مهندسی پروتئین، اشاره به اصلاح توالی پروتئین با استفاده از روشهای ژنتیکی دارد. تکنیک های مهندسی پروتئین جهت
1) شناسایی مکانیزمهای آنزیم،
2) تغییر محل اتصال آنزیم یا آنتی بادی به صورت ارادی،
3) تغییر خواص کلی آنزیم، مانند پایداری آنزیم در برابر دمای بالا، pH شدید، پروتئازها، حلالیت آن یا خاصیت آنتی ژنی آن انجام میگیرد.
اگر یک ساختار پروتئینی شناخته شده به عنوان نقطه آغازگر استفاده شود و اسیدهای آمینه یا توالی های منحصر به فرد با محلهای موتاژنز هدفمند اختصاصی جایگزین شوند، که به آن پروتکل طراحی پروتئینی هوشمندگفته می شود. تبادل ژنتیکی اسید آمینه به طور تصادفی و انتخاب بازده توسط خواص بهبود یافته خود، تکامل هدایت شده خوانده میشود.
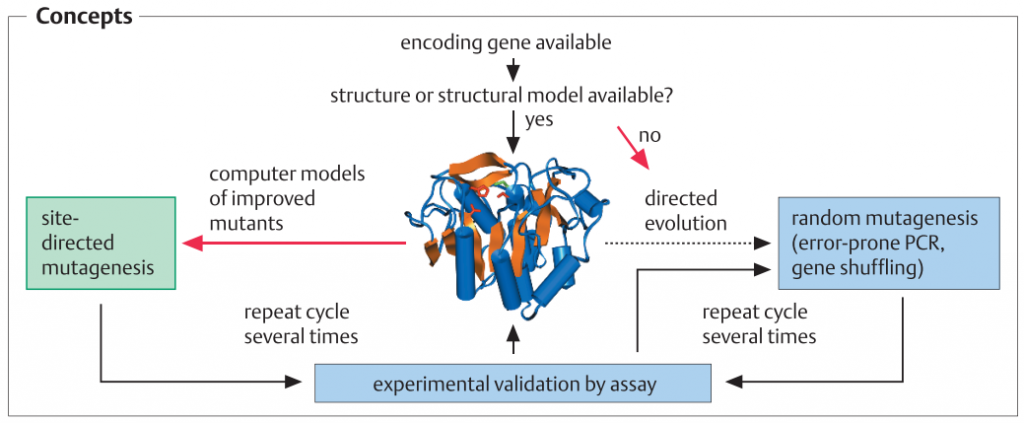
متد های کلی طراحی پروتئین یا مهندسی پروتئین :
هر دو طراحی پروتئین هوشمند و تکامل هدایت شده، نیاز به ژن کدگذاری پروتئین دارند. برای طراحی پروتئین هوشمند، اطلاعات ساختاری در مورد پروتئین مورد نیاز است؛ برای آن می توان از ساختار اشعه ایکس، از داده های ساختاری NMR یا از یک مدل ساختاری حاصل از ساختار سوم پروتئین های مرتبط و هماهنگ با مدل سازی همولوگ استفاده کرد.پ
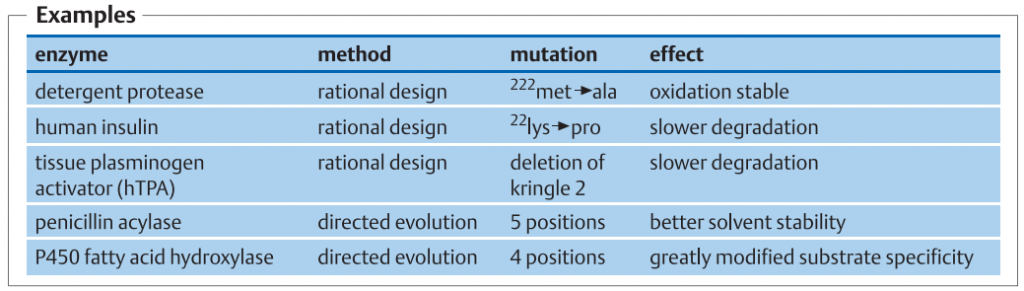
موتاژنز طراحی پروتئین یا مهندسی پروتئین :
در طراحی پروتئین های هوشمند، اسید آمینه های منحصربه فرد مبادله، یا یک توالی آمینواسیدی معرفی یا حذف می شود. این فرآیند در سطح DNA و توسط PCR انجام می شود. پروتکل های متعددی در دسترس هستند که انجام این تغییرات را با روش های سریع، ساده و قابل اطمینان امکان پذیر می سازد. برای موتاژنز تصادفی، ژن را می توان در یک سلول میزبان E. coli کلون کرد که مکانیزم های اصلاح DNA آن مختل شده و تحت شرایط جهشزا کشت داده می شود. متعاقبا ، یک پروتکل PCR به کار گرفته می شود که در آن با اضافه کردن یون های Mn2+ یا مواد دیگر باعث افزایش تعداد اشتباهات ساختگی در طول افزایشDNA می شود (1-3%). روش جابجایی ژن[1]، یکی دیگر از روش هایست که بر اساس ایجاد یک کتابخانه قطعات DNA از ژن های مرتبط (تطابق توالی آنها حدود 80٪) و ترکیب مجدد قطعات با روش PCR، و سپس غربالگری با توان بالا برای خواص مطلوب انجام می گیرد.
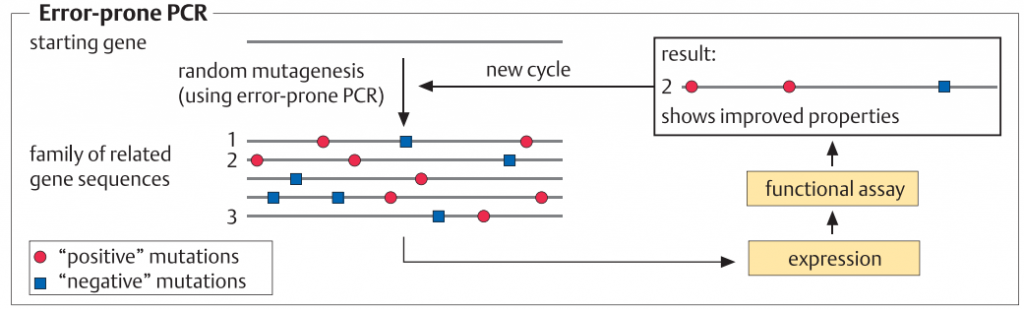
طراحی پروتئین هوشمند
ساختار سوم پروتئین معمولا توسط کریستالوگرافی اشعه ایکس و گاهی توسط تکنیک های NMR سه بعدی از پروتئین های 13C- و 15N- حاصل می شود. از سال 2014، مختصات بیش از ،000 100ساختار پروتئین در ProteinDatabase (PDB) موجود است که از طریق اینترنت به شکل بین المللی قابل دسترسی هستند. اگر توالی پروتئین مد نظر، بیش از 30٪ همولوژی با پروتئینی که مختصات آن در دسترس است نشان دهد، مدل سازی همولوژی از ساختمان ناشناخته ای که بر اساس مختصات شناخته شده انجام شده یک مدل ساختاری از پروتئین ناشناخته را فراهم میکند که برای آزمایشات جهش زایی به اندازه کافی دقیق است.
تا کنون، با توجه به قدرت محدود کامپیوتر، چنین شبیه سازی تنها در خلاء امکان پذیر بود. با ظهور ابرکامپيوترها و کامپيوترهاي بسيار موازي، مدلسازي ساختار پروتئين، پروتئين هاي جهش یافته و اتصال آنها به سوبسترا يا آنتي ژنها ميتواند در حلال انجام شود (براي اين منظور ممکن است محاسبات مکانیک مولکولی (محاسبات نیروی میدان) از تعاملات چند ده تا هزاران اتم مورد نیاز باشد!). با وجود پیشرفت، پیش بینی هایی که از این روش ها در روش های نرم افزاری حاصل می شود، بایستی معمولا با چندین دوره شبیه سازی و آزمایشهای ژنتیکی (چرخه های مهندسی پروتئین) بهینه شوند. طراحی پروتئین اغلب با جایگزینی همه اسیدهای آمینه پروتئین زا در محل هایی، همراه است که با جای خالی سوبسترا یا با توجه به بررسی های آنالوژِی به عنوان محلهای مربوطه، برای اتصال یک سوبسترا (اشباع موتاژنز) انتخاب شدهاند.
تکامل هدایت شده طراحی پروتئین یا مهندسی پروتئین
در این روش بر خلاف طراحی پروتئین هوشمند، مدل های ساختاری برای این تکنیک ضروری نیستند. برای بهینه سازی اتصال آنتی بادیهای انتخابی، تکنیک نمایش فاژ (phage display) به طور موثر مورد استفاده قرار گرفته است: این اجازه می دهد تا کتابخانه های بزرگ از آنتی بادی های جهش یافته (تا 10 10) غربالگری شوند. در مورد آنزیم ها، ژن کد گذار تحت جهش زایی تصادفی قرار می گیرد، ژنهای جهش یافته در یک کتابخانه جهش یافته بیان می شوند و جهش ها برای خواص مد نظر مورد بررسی قرار می گیرند.
با برنامه های جهش زایی اشباع مکرر، رویکردهای هدفمندتر برای بررسی توالی پروتئین بزرگ به سرعت در دسترس قرار می گیرد. بررسی کیفیت آنزیم از اهمیت بسیار زیادی برخوردار است چرا که سرعت ایجاد و همچنین کیفیت جهش را تعیین می کند. در سال های اخیر، این روش نتایج خوبی برای توسعه آنزیم های صنعتی به ارمغان آورده است. برای مثال در تغییر سوبسترای اختصاصی آنها یا ثبات ترمو- و آلکالی آنها. سیستم های رباتیک یا تجهیزات FACS[2] برای این روش ها مورد استفاده قرار گرفته است.
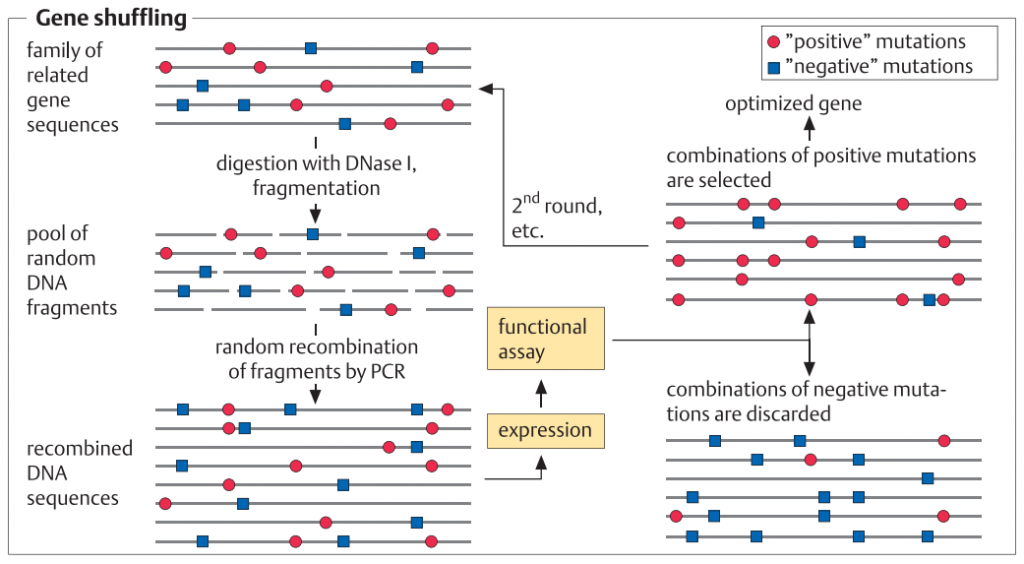
[1] Gene shuffling
[2] flow-activated cell sorters
نویسنده : ساناز فرخی – دانشجو ارشد بیوتکنولوژی میکروبی




1 نظر
برنامه های جهش زایی اشباع مکرر خیلی مبهم بودو ایا برای طراحی CAR T CEll هم همانند مورد آنتی بادی از تکنیک نمایش فاژی استفاده میشود؟ چطور؟ آیا طراحی CAR Tcell نیازمند مهندسی پروتئین است ؟ در کدام بخش ها میتوان تغییرات دلخواه را ایجاد کرد؟ با چه روشی؟