واکنش زنجیرهای پلیمراز (به انگلیسی: Polymerase Chain Reaction) که مخفف آن PCR میباشد. واکنش زنجیرهای پلیمراز (PCR) تکنیکی در زیستشناسی مولکولی است و به منظور تکثیر یک نسخه منفرد یا نسخههای کمی از یک قطعه DNA با توالی خاص به تعداد هزار یا میلیونها نسخه به کار میرود. این تکنیک ابزاری آسان و ارزان قیمت برای تکثیر یک قطعه خاص از DNA است و برای اهدافی همچون تشخیص و نظارت بر بیماریهای ژنتیکی، شناسایی مجرمان (در زمینهٔ پزشکی قانونی) و مطالعه عملکرد یک بخش هدف از DNA مورد استفاده قرار میگیرد.[۱]
واکنش زنجیرهای پلیمراز
این تکنیک در سال 1983 بوسیله کری مالیس ابداع شد[۲][۳]). هماکنون PCR یک تکنیک متداول و اغلب ضروری در آزمایشگاههای بالینی و آزمایشگاههای تحقیقاتی است و در موارد گوناگونی کاربرد دارد.[۴][۵] این کاربردها شامل کلونکردن DNA برای توالی یابی، فیلوژنی بر پایه DNA، آنالیز عملکرد ژنها، تشخیص بیماریهای ارثی، شناسایی اثر انگشت ژنتیکی (مورد استفاده در علم پزشکی قانونی) و تشخیص عوامل بیماریزا در تستهای نوکلئیک اسید برای تشخیص بیماریهای عفونی است. در سال ۱۹۹۳ مولیس همراه با مایکل اسمیت جایزه نوبل شیمی را برای کار روی PCR دریافت کردند.[۶]
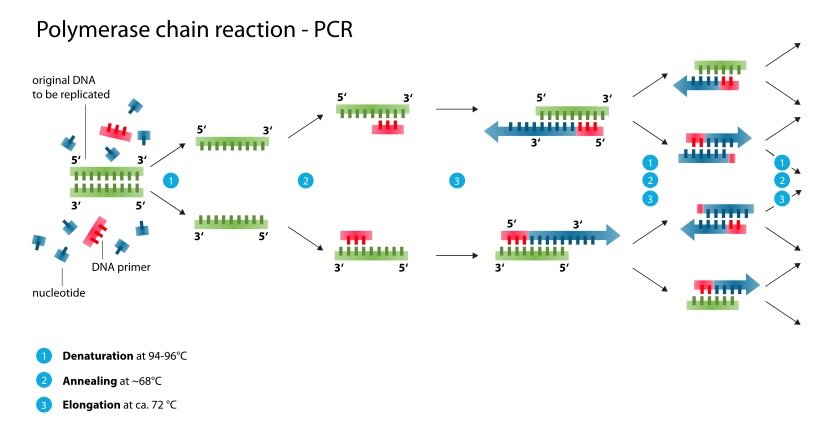
اساس تکنیک PCR چرخههای حرارتی است. این چرخهها شامل چرخههای گرمایی و سرمایی تکراری، ذوب DNA و تکثیر آنزیمی DNA است. پرایمرها (قطعات کوتاه DNA) که حاوی توالی مکمل ناحیه هدف اند به همراه یک DNA پلیمراز، اجزای اصلی واکنش PCR برای انتخاب و تکثیر قطعه مورد نظر را تشکیل میدهند. طی فرایند PCR الگوی DNA به صورت لگاریتمی تکثیر میشود و DNA تکثیر شده خود به عنوان الگویی برای همانندسازی استفاده میشود. PCR میتواند به شکل گستردهای برای انجام مراحل مختلف در دستکاریهای ژنتیکی استفاده شود.
تقریباً در تمام کاربردهای PCR از یک DNA پلیمراز مقاوم به حرارت مانند تک پلیمراز (آنزیمی که از باکتری Thermus aquaticus جداسازی شده) استفاده میشود. این DNA پلیمراز از یک DNAی تکرشتهای به عنوان الگو استفاده کرده و با کمک پرایمرها و با بهکارگیری نوکلئوتیدها که بلوکهای ساختاری DNA هستند، یک رشته DNAی جدید میسازد. در اغلب روشهای PCR از چرخههای حرارتی یعنی گرم و سرد کردن متناوب نمونههای PCR طبق مراحل دمایی مشخص استفاده میشود.
در مرحله اول، دو رشتهٔ مارپیچ DNA دورشتهای در یک دمای بالا، در فرآیندی که ذوب DNA نامیده میشود از یکدیگر جدا میشوند. در مرحله دوم، دما پایین آورده شده و و هریک از دورشته DNA به عنوان الگو عمل میکنند. ساخته شدن رشته جدید از روی الگو توسط DNA پلیمراز انجام میشود. پرایمرها بر اساس توالی قطعهای از DNA که موردنظر ماست طراحی میشوند.
اصول
PCR یک ناحیه خاص از رشته DNA هدف را تکثیر میکند. بهطور معمول اکثر روشهای PCR این قابلیت را دارند تا قطعات DNA بین ۱/۰ و ۱۰ کیلوجفت باز (kbp) را تکثیر کنند. هرچند تکنیکهای دیگر میتوانند قطعاتی با اندازههای بالاتر از ۴۰ کیلوجفت باز را نیز تکثیر کنند.[۷] مقدار تکثیر محصول بوسیله سوبسترای موجود در واکنش که پیشرفت واکنش را محدود میکند تعیین میشود.[۸]
یک واکنش PCR پایهای نیازمند چندین جزء است.[۹] این اجزاء شامل موارد زیر است:
دی ان ای الگو که حاوی ناحیه DNA ی هدف برای تکثیر است.
تگ پلیمراز که یک DNA پلیمراز مقاوم به حرارت است[۱۰]
دو پرایمر DNA که مکمل انتهای ۳’ رشتههای سنس و آنتی سنس DNA ی الگو هستند (بدون پرایمرها، جایگاه آغاز دورشتهای که DNA پلیمراز بتواند به آن متصل شود شناخته نمیشود).[۱۱] پرایمرهای خاصی که مکمل DNA هدف هستند از قبل انتخاب شده و به شکل سفارشی در آزمایشگاه ساخته یا از تأمین کنندگان خریداری میشوند.
دزوکسی نوکلئوتیدهای سه فسفاته یا dNTPs بلوکهای ساختاری هستند که DNA پلیمراز با استفاده از آنها رشتههای جدید را میسازد.
یک محلول بافری که محیط شیمیایی مناسبی برای بهبود فعالیت و پایداری DNA پلیمراز فراهم میکند.
کاتیونهای دوظرفیتی مانند منیزیوم (Mg) یا منگنز (Mn)؛ کاتیون Mg2+ متداولتر است. همچنین کاتیون Mn2+ میتواند برای جهش زایی DNA ی حاصل از PCR استفاده شود و غلظت بالای این یون نرخ خطا را در طول سنتز افزایش میدهد.[۱۲]
کاتیونهای تک ظرفیتی، معمولاً یونهای پتاسیم (K)
بافر 10X
واکنش معمولاً در حجم ۱۰ تا ۲۰۰ میکرولیتر در لولههای کوچک واکنش (با حجم ۲/۰–۵/۰ میلی لیتر) و در یک چرخه حرارتی انجام میشود. چرخههای حرارتی لولههای واکنش را به منظور فراهم کردن دمای لازم در هر مرحله سرد و گرم میکنند. بسیاری از چرخههای حرارتی پیشرفته از اثر پلتیر Peltier effect که اجازه گرم و سرد کردن لولههای PCR را بوسیله معکوس کردن جریان الکتریکی میدهد استفاده میکنند.
روش
بهطور کلی PCR شامل مجموعهای از ۴۰–۲۰ بار تغییر دمایی تکرارشونده به نام سیکل است که هر سیکل متداولاً از دو یا سه مرحله دمایی مستقل تشکیل شدهاست.[۱۳] مراحل مشترک اغلب روشهای PCR عبارت است از:
نشانگر ژنتیکی یک جاندار با استفاده از واکنشزنجیرهایپلیمراز
آغاز: این مرحله برای DNA پلیمرازی که نیازمند فعالسازی گرمایی بوسیله Hot-start PCR است[۱۴] لازم میباشد و شامل گرم کردن محفظه واکنش تا دمای ۹۶–۹۴ درجه سانتی گراد (۲۰۵–۲۰۱ درجه فارنهایت) یا ۹۸ درجه سانتی گراد (۲۰۸ درجه فارنهایت) برای پلیمرازهای بسیار مقاوم به حرارت است. این مرحله ۱۰–۱ دقیقه به طول میانجامد.
دناتوراسیون: این مرحله اولین مرحله سیکل است و شامل گرم کردن محفظه واکنش تا ۹۸–۹۴ درجه سانتی گراد (۲۰۸–۲۰۱ درجه فارنهایت) به مدت ۳۰–۲۰ ثانیه میشود که بوسیله شکستن پیوندهای دورشتهای بین بازهای مکمل باعث ذوب شدن یا دناتوراسیون DNA الگوی دورشتهای شده و به این ترتیب دو مولکول DNA تک رشتهای حاصل میشود.
اتصال: در این مرحله دمای واکنش به مدت ۴۰–۲۰ ثانیه به ۶۵–۵۰ درجه سانتی گراد کاهش مییابد که باعث میشود پرایمرها به هریک از الگوهای تک رشته DNA متصل شوند. اتصال معمولاً حدود ۵–۳ درجه سانتی گراد زیر دمای ذوب(Tm پرایمرها انجام میشود. در طول این فرایند DNA پلیمراز به ترکیبی از الگو و پرایمر متصل و شروع به ایجاد رشتههای جدید میکند).
گسترش/ طویل شدن: در این مرحله دما برای عملکرد DNA پلیمراز لازم است؛ دمای بهینه یک DNA پلیمراز ۸۰–۷۵ درجه سانتی گراد است. با این وجود معمولاً دمای ۷۲ درجه سانتی گراد برای این آنزیم استفاده میشود[۱۵][۱۶]). در این مرحله DNA پلیمراز یک رشته DNA ی جدید که مکمل رشته الگو است را در جهت ’۵ به ’۳ سنتز میکند. زمان لازم برای افزایش طول بستگی به DNA پلیمراز استفاده شده و طول ناحیه DNA ی هدف برای تکثیر دارد.
فرآیندهای دناتوراسیون، اتصال و طویل شدن یک سیکل را تشکیل میدهند. برای تکثیر DNA ی هدف تا میلیونها نسخه، سیکلهای متعددی نیاز است.
طویل شدن نهایی: این تک مرحله اختیاری است و پس از آخرین سیکل PCR برای اطمینان از طویل شدن کامل هر تک رشته DNA در یک دمای ۷۴–۷۰ درجه سانتی گراد (۱۶۵–۱۵۸ درجه فارنهایت) به مدت ۱۵–۵ دقیقه انجام میشود.
نگهداری نهایی: مرحله نهایی سرد کردن محفظه واکنش تا ۱۵–۴ درجه سانتی گراد برای یک زمان نامحدود است و ممکن است این مرحله برای ذخیرهسازی کوتاه مدت محصولات PCR استفاده شود.
به منظور بررسی میزان موفقیت PCR انجام شده از الکتروفورز بر روی ژل (به منظور جداسازی محصولات PCR و بررسی اندازه قطعات استفاده میشود.
در طراحی پرایمربرای واکنش PCR باید نکات خاصی در نظر گرفته شود مثلاً حتماً قسمت ۳پریم از پرایمر میبایست مکمل بخش هدف باشد که این برای بخش ۵ پریم الزامی نمیباشد همچنین باید در نظر گرفته شود که دو پرایمر حداقل توالی مکمل یکدیگر را داشته باشند و توالیهای مستعد تشکیل سنجاق سری هم در آنها تا حد معمول استفاده نشود. معمولاً از گرم کردن و سرد کردن مخلوط واکنش برای جدا شدن دو رشته DNA و چسبیدن آغازگرها یا Annealing استفاده میشود. در نتیجه این چرخهها میلیونها نسخه از قطعه کوتاهی از DNA تولید میشود.
منابع
- ↑ 1. “PCR”. Genetic Science Learning Center, University of Utah.
- ↑ 2. Jump up^ Bartlett, J. M. S. ; Stirling, D. (2003). “A Short History of the Polymerase Chain Reaction”. PCR Protocols. Methods in Molecular Biology. 226 (2nd ed.). pp. 3–6. doi:10.1385/1-59259-384-4:3. شابک ۱-۵۹۲۵۹-۳۸۴-۴.
- ↑ 3. Jump up^ Mullis, Kary B. et al. “Process for amplifying, detecting, and/or-cloning nucleic acid sequences” U.S. Patent 4,683,195
- ↑ 4. ^ Jump up to:a b c Saiki, R. ; Scharf, S. ; Faloona, F. ; Mullis, K. ; Horn, G. ; Erlich, H. ; Arnheim, N. (1985). “Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia”. Science. 230 (4732): 1350–1354. doi:10.1126/science.2999980. PMID 2999980.
- ↑ 5. ^ Jump up to:a b Saiki, R. ; Gelfand, D. ; Stoffel, S. ; Scharf, S. ; Higuchi, R. ; Horn, G. ; Mullis, K. ; Erlich, H. (1988). “Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase”. Science. 239 (4839): 487–491. doi:10.1126/science.2448875. PMID 2448875
- ↑ 6. ^ Jump up to:a b “Kary B. Mullis – Nobel Lecture: The Polymerase Chain Reaction”.
- ↑ 7. Jump up^ Cheng, S. ; Fockler, C. ; Barnes, W. M. ; Higuchi, R. (1994). “Effective Amplification of Long Targets from Cloned Inserts and Human Genomic DNA”. Proceedings of the National Academy of Sciences. 91 (12): 5695–5699. doi:10.1073/pnas.91.12.5695. PMC 44063. PMID 8202550.
- ↑ 8. Jump up^ Carr AC, Moore SD (2012). Lucia, Alejandro, ed. “Robust quantification of polymerase chain reactions using global fitting”. PLoS ONE. 7 (5): e37640. doi:10.1371/journal.pone.0037640. PMC 3365123. PMID 22701526.
- ↑ 9. ^ Jump up to:a b Joseph Sambrook & David W. Russel (2001). Molecular Cloning: A Laboratory Manual (3rd ed.). Cold Spring Harbor, N.Y. : Cold Spring Harbor Laboratory Press. ISBN 0-879-69576-5. Chapter 8: In vitro Amplification of DNA by the Polymerase Chain Reaction
- ↑ 10. Jump up^ “Polymerase Chain Reaction (PCR)”. National Center for Biotechnology Information, U.S. National Library of Medicine.
- ↑ “PCR”. Genetic Science Learning Center, University of Utah.
- ↑ 11. Jump up^ Pavlov, A. R. ; Pavlova, N. V. ; Kozyavkin, S. A. ; Slesarev, A. I. (2004). “Recent developments in the optimization of thermostable DNA polymerases for efficient applications☆”. Trends in Biotechnology. 22 (5): 253–260. doi:10.1016/j.tibtech.2004.02.011. PMID 15109812.
- ↑ 12. Jump up^ Rychlik W, Spencer WJ, Rhoads RE (1990). “Optimization of the annealing temperature for DNA amplification in vitro”. Nucl Acids Res. 18 (21): 6409–6412. doi:10.1093/nar/18.21.6409. PMC 332522. PMID 2243783.
- ↑ 13. ^ Jump up to:a b Sharkey, D. J. ; Scalice, E. R. ; Christy, K. G. ; Atwood, S. M. ; Daiss, J. L. (1994). “Antibodies as Thermolabile Switches: High Temperature Triggering for the Polymerasehttps://upload.wikimedia.org/wikipedia/fa/4/4a/Button_numbers.png Chain Reaction”. Bio/Technology. 12 (5): 506–509. doi:10.1038/nbt0594-506.
- ↑ 14. ^ Jump up to:a b Chien A, Edgar DB, Trela JM (1976). “Deoxyribonucleic acid polymerase from the extreme thermophile Thermus aquaticus”. J. Bacteriol. 127 (3): 1550–1557. PMC 232952. PMID 8432.
- ↑ 15. ^ Jump up to:a b Lawyer, F. ; Stoffel, S. ; Saiki, R. ; Chang, S. ; Landre, P. ; Abramson, R. ; Gelfand, D. (1993). “High-level expression, purification, and enzymatic characterization of full-length Thermus aquaticus DNA polymerase and a truncated form deficient in 5′ to 3′ exonuclease activity”. PCR methods and applications. 2 (4): 275–287. doi:10.1101/gr.2.4.275. PMID 8324500.


