تکنیکهای توالی یابی | sequencing | توالی یابی نسل جدید
بسیاری از متدهای امروزی دستکاری DNA، RNA و پروتئینها، وابسته به وجود دانش قبلی راجع به توالی نوکلئوتیدی ژنوم موردنظر هستند. اما خود این توالیها برای نخستین بار چگونه شناخته شدهاند؟ و امروزه توالی مولکولهای DNA و RNA چگونه تعیین میگردد؟ تکنیک توالی یابی (sequencing) فرایند تعیین ترتیب دقیق بازها در نوکلئیکاسیدها است و باعث جهش ناگهانی در دانش ما از ژنتیک شده است. روشهای انجام هر چه سریعتر و آسانتر این فرایند عامل پیشبرنده پیشرفتهای اخیر بیوتکنولوژی است.
ایجاد کتابخانههای DNA پایه تکنیکهای توالی یابی ژنوم بود و انجام آن را امکان پذیر ساخت. در اواخر دهه ۱۹۷۰ بود که محققان تکنیکهایی را برای تعیین سریع و آسان توالیهای نوکلئوتیدی قطعات DNA خالص معرفی کردند. متدی که تاکنون بیشتر از همه مورد استفاده قرار گرفته است، تکینک توالی یابی سنگر نام دارد و اساس آن آنزیمی میباشد. طی این متد DNA پلیمراز با استفاده از توالیهای الگوی کلون شده و تکرشتهای، رشته جدید را سنتز میکند. توالی یابی Sanger زیربنای تمام تکنولوژيهای توالی یابی است که امروزه مورد استفاده قرار میگیرند و ژنومیکس (علم مطالعه ژنوم) را شکل داده است.
تکنیک سنگر ( Sanger )
تکنیک Sanger به منظور تعیین توالی ژنومهای فراوانی مانند E. coli، مگس سرکه، کرمهای نواری (نماتودها)، موش و انسان مورد استفاده قرار گرفته است. Sanger و همکارانش نخستین دانشمندانی بودند که کل ژنوم یک جاندار را با استفاده از این تکنیک توالی یابی کردند. این ژنوم طولی در حدود ۵۰۰۰ باز داشت و متعلق به ویروسی به نام phiX174 بود. پس از این دستاورد آنان به تعیین توالی ژنومهای دیگری مانند ژنوم میتوکندری انسان ادامه دادند.
توالی یابی DNA با استفاده از تکنیک Sanger در توالی یابی ابتدایی ژنوم انسان بسیار کمک کننده بود. با این حال متدهای ابتدایی آهسته بودند، نیاز به نیروی انسانی فراوانی داشتند، هزینه تهیه مواد اولیه بالا بود، پیشرفت واکنشها نیاز به شرایط پیچیدهای داشت و بازده واکنشها نیز پایین بود. در نتیجه سالها طول میکشید که یک یا دو ژن توالی یابی شوند.
توالی یابی ژنوم انسان
توالی مرجع ژنوم انسانی برای نخستین بار در سال ۲۰۰۳، با صرف یک میلیارد دلار هزینه و در طول ۱۳ سال تکمیل شد. در طول این پروژه هزینه تعیین توالی هر باز به علت وقوع پیشرفت در متد خاتمه زنجیر به همراه الکتروفورز موئین کاهش پیدا کرد و باعث شد که به بودجهای کمتر از حد انتظار نیاز داشته باشد. هزینه توالی یابی برای یک میلیون باز DNA در سپتامبر ۲۰۰۱، ۵۲۹۲ دلار و برای کل ژنوم انسان ۹۵ میلیون دلار بود.
در طی دهه اخیر، تکنولوژیهای توالی یابی پیشرفت فراوانی کردهاند؛ به نحوی که میتوان در عرض چند ساعت و با صرف هزینه کمتر، کل ژنوم انسان را که دارای طولی در حدود ۳.۲ بیلیون باز است، توالی یابی کرد. به طوری که تا اکتبر ۲۰۱۳ هزینه توالی یابی یک میلیون جفت باز به کمتر از ۶ سنت و درنتیجه هزینه توالی یابی کل ژنوم به حدود ۵۰۹۶ دلار رسید.
علت وقوع این کاهش چشمگیر، تکنیکهای توالی یابی نسل جدید (Next generation sequencing) بودند. معرفی توالی یابی نسل جدید در سال ۲۰۰۷ هیجانی در علم ژنومیکس پدید آورد و روشی کاملا جدید برای توالی یابی معرفی نمود. علاوه بر کاهش زمان و هزینه موردنیاز، تعداد ژنومهای تعیین توالی شده نیز با ابداع این تکنیکها افزایش قابل توجهی پیدا کرد. از جمله این متدها میتوان به تکنیک توالی یابی ۴۵۴ و Illumina اشاره کرد.
امروزه حتی متدهای جدیدتر، دقیقتر، ارزانتر و سریعتری نیز در حال توسعه هستند و تکنولوژیهای نسل سوم خوانده میشوند. این تکنیکها فاقد تمام مراحل تکثیر DNA هستند و توالی یک مولکول واحد DNA را تعیین میکنند. یکی از جدیدترین تکنیکهای توالی یابی نسل سوم، SMRT نام دارد و از جمله ویژگیهای خارقالعاده آن، کاهش نیاز به مواد اولیه گران قیمت، حساسیت بالا و پدید آوردن امکان مشاهده DNA پلیمراز هنگام ساخت رشته جدید است. استفاده از این تکنیک برای توالی یابی ژنوم انسان هنوز امکانپذیر نیست، اما اگر ممکن شود، خواهیم توانست به کمک آن کل ژنوم را در حدود یک ساعت توالی یابی کنیم. به احتمال زیاد در آینده شاهد توسعه متدهایی که حتی سریعتر و ارزانتر از متدهای کنونی هستند نیز خواهیم بود.
- تکنیک توالی یابی Sanger
- تکنیک توالی یابی نسل جدید
- تکنیک توالی یابی ۴۵۴
- تکنیک توالی یابی Illumina
- تکنیک توالی یابی نسل سوم
-
تکنولوژی توالی یابی SOLID یا oligonucleotide Ligation and detection
تکنیک توالی یابی سنگر ( Sanger )
در سال ۱۹۷۴ Fredrick Sanger تکنیکی را به منظور تعیین توالی ژنها در محیط in vitro ابداع کرد. او به توالی آمینواسیدی انسولین علاقهمند بود و تصمیم گرفت که با استفاده از توالی نوکلئوتیدی آن به توالی آمینواسیدیاش برسد. متدی که Sanger و همکارانش در Medical Research Council Laboratory کمبریج اختراع کردند، روش خاتمه زنجیره سازی (Chain termination) نام گرفت و امروزه نیز کاربرد دارد. از جمله علل محبوبیت این تکنیک اتوماسیون آسان آن است.
توالی یابی سنگر ( Sanger )
توالی یابی Sanger همانند فرایند تکثیر DNA نیاز به پرایمر (رشته الیگونوکلئوتیدی کوتاهی از جنس RNA) که باید به نقطه یکسانی در تمام مولکولها متصل گردد)، DNA پلیمراز، توالی الگوی تکرشتهای و دئوکسی نوکلئوتیدها دارد. در طی واکنش، این مواد در محیط in vitro با یکدیگر مخلوط میشوند و پلیمراز نسخههای فراوانی را از توالی اصلی تولید میکند. اساس این تکنیک فرایند طبیعی همانندسازی DNA است و واکنش در حالت عادی میتواند تا پلیمریزه شدن هزاران نوکلئوتید ادامه پیدا کند؛ با این تفاوت که در این مورد همانندسازی در نقاطی قطع می شود و رشتههایی در اندازههای متفاوت به دست میآیند.
سوبسترای واکنش غالبا DNA نوترکیبی بود که در اثر دناتوراسیون توانایی اتصال به پرایمر توالییابی اختصاصی رشته را پیدا میکرد. قطعات DNA در وکتورهای فاژمیدی که در اثر دستکاری میتوانستند DNAهای نوترکیب تکرشتهای تولید کنند، نیز کلون میشدند. روش جایگزینی که امروزه نیز کاربرد فراوانی دارد، تولید قطعات DNA الگو با استفاده از PCR و تبدیل مولکولهای حاصل به فرم تک رشتهای است. در این حالت پلیمراز مورد استفاده نباید دارای خاصیت proofreading باشد تا سرعت الحاق نوکلئوتیدها افزایش یابد. محصول نهایی در تمام موارد ذکرشده، نسخههای فراوانی از مولکول DNA تکرشتهای موردنظر است.
نخستین ترفند به دست آوردن توالی، متوقف کردن سنتز رشته جدید در هر کدام از جفتبازها است. این کار با مهار تصادفی سنتز زنجیره جدید DNA و درنتیجه تولید رشتههایی با طولهای متفاوت که میتوانند بر مبنای اندازه از یکدیگر جدا شوند، صورت میگیرد. در نتیجه تفاوت اندازه قطعات تولیدشده با یکدیگر در حدود یک جفت باز خواهد بود و در الکتروفورز نردبانی از قطعات به دست خواهد آمد. ترفند دوم تعیین ماهیت نوکلئوتید آخر هر قطعه است که اگر مشخص باشد، میتوان توالی را به طور مستقیم از روی ژل خواند. اما سوال این است که چگونه میتوان باز آخر هر قطعه را در نردبان توالی یابی تعیین کرد؟
DNA پلیمراز سنتز رشته جدید DNA را بر اساس توالی رشته الگو انجام میدهد. زنجیره DNA حاوی دئوکسی نوکلئوتیدهایی است که دارای گروه هیدروکسیل در موقعیت ۳’ حلقه دئوکسی ریبوز میباشند. DNA پلیمراز افزودن نوکلئوتید بعدی را با اتصال فسفات روی کربن ۵’ آن، به ۳’-هیدروکسیل نوکلئوتید پیشین و با تشکیل پیوند فسفودیاستر انجام میدهد. در صورتیکه یکی از نوکلئوتیدها فاقد ۳’-هیدروکسیل باشد، نوکلئوتید دیگری افزوده نخواهد شد و زنجیره به طور ناگهانی خاتمه خواهد یافت.
در حین واکنش توالی یابی سنگر، درصد معینی از نوکلئوتیدها که فاقد ۳’-هیدروکسیل هستند و دی دئوکسی نوکلئوتید (dideoxynucleotide: ddNTP) نامیده میشوند، با دئوکسی نوکلئوتیدهای نرمال مخلوط میشوند. آنزیمهای پلیمراز توانایی افتراق دئوکسی نوکلئوتیدها از دی دئوکسی نوکلئوتیدها را ندارند و این نوع از نوکلئوتیدها در صورت اتصال، به عنوان خاتمه دهنده زنجیره اختصاصی باز (base specific chain terminator) عمل خواهند کرد.
همان طور که گفته شد دئوکسی ریبونوکلئوتیدها در مقادیر بسیار بالاتری نسبت به دی دئوکسی ریبونوکلئوتیدها حضور دارند، در نتیجه خاتمه سنتز زنجیره جدید همواره در نزدیکی پرایمر رخ نمیدهد. در واقع ممکن است پلیمریزاسیون چند صد نوکلئوتید صورت بگیرد، پیش از آن که باز خاتمه دهنده رشته افزوده شود. در چنین واکنشهایی معمولا حداکثر طول قطعات ۸۰۰ نوکلئوتید خواهد بود.
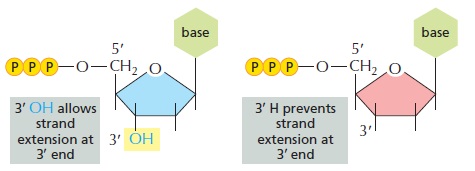 تصویر ۱ . در دی دئوکسی ریبونوکلئوتیدها، گروه هیدروکسیلی که در نوکلئوتیدهای نرمال به کربن ۳’ متصل است، با اتم هیدروژن جایگزین میگردد.
تصویر ۱ . در دی دئوکسی ریبونوکلئوتیدها، گروه هیدروکسیلی که در نوکلئوتیدهای نرمال به کربن ۳’ متصل است، با اتم هیدروژن جایگزین میگردد.
تکنیک توالی یابی Sanger بر این پدیده استوار است که مولکولهای DNA تکرشتهای که تفاوتشان با یکدیگر تنها در حد یک نوکلئوتید است، میتوانند توسط الکتروفورز در ژل پلی آکریل آمید از یکدیگر جدا شوند. اساس این نوع از الکتروفورز با الکتروفورز در ژل آگارز یکسان است، با این تفاوت که حفرات در ژل پلی آکریل آمید کوچکتر هستند و در نتیجه امکان تفکیک قطعات کوچکتر را با دقت بالاتری پدید میآورند. در صورتی که این نوع از الکتروفورز از نوع Denaturing باشد، به علت وجود مقادیر بالای اوره و سایر ترکیبات دناتورهکننده مولکولهای DNA به صورت تکرشتهای باقی خواهند ماند.
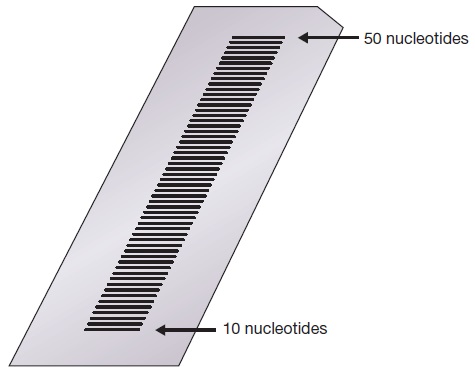 ۲ . الکتروفورز در ژل پلی آکریل آمید مولکولهای تکرشتهای DNA که تنها در یک نوکلئوتید با همدیگر تفاوت دارند را از همدیگر تفکیک کنند. مولکولهای DNA با مارکر رادیواکتیو لیبل شده اند و مشاهده آنها با اتورادیوگرافی صورت میگیرد. در اثر قرار گرفتن فیلم در معرض اشعه X، DNAهای لیبل شده به رنگ سیاه در میآیند.
۲ . الکتروفورز در ژل پلی آکریل آمید مولکولهای تکرشتهای DNA که تنها در یک نوکلئوتید با همدیگر تفاوت دارند را از همدیگر تفکیک کنند. مولکولهای DNA با مارکر رادیواکتیو لیبل شده اند و مشاهده آنها با اتورادیوگرافی صورت میگیرد. در اثر قرار گرفتن فیلم در معرض اشعه X، DNAهای لیبل شده به رنگ سیاه در میآیند.
نحوه خواندن توالی یابی سنگر
نحوه خواندن توالی از پایین به بالا است؛ چون قطعاتی که نزدیک پرایمر خاتمه یافتهاندو سریعتر از مولکولهایی حرکت میکنند که در نقطهای دور از پرایمر جدا شده اند. نوارهای الکتروفورز به صورت نردبانی مشاهده میشوند که پلههای آن به اندازه یک نوکلئوتید از هم فاصله دارند. درنتیجه هر نوار نشانگر نوکلئوتیدی با توالی مکمل دی دئوکسی نوکلئوتیدی است که زنجیره را در آن نقطه خاتمه داده است. به عنوان مثال اگر دی دئوکسی نوکلئوتید A زنجیره را خاتمه داده باشد، نوکلئوتید موجود در رشته اصلی T است.
توالی یابی Sanger به صورت چهار واکنش موازی انجام میگیرد که هر کدام شامل ۴ نوع دئوکسی ریبونوکلئوتید (dATP، dCTP، dGTP، dTTP) به همراه درصد کمی از یکی از انواع دی دئوکسی نوکلئوتید میباشد. یکی از ۴ دئوکسی نوکلئوتید و یا پرایمر باید لیبل شده باشند تا رشته در حال سنتز نیزعلامتگذاری شود. با تنظیم غلظت دی دئوکسی نوکلئوتید در حدی بسیار پایینتر از آنالوگ دئوکسی نوکلئوتیدی آن، رقابتی بین این دو به منظور اتصال به رشته DNA در حال ساخت شکل خواهد گرفت. مقدار دئوکسی نوکلئوتید بسیار بیشتر است و با اتصال آن، سنتز DNA ادامه پیدا میکند، تا اینکه گاهی اوقات ناگهان با اتصال دی دئوکسی نوکلئوتید، پلیمریزاسیون متوقف و تولید زنجیره خاتمه مییابد.
از آن جایی که نمونه DNA شامل جمعیتی از مولکولهای یکسان است، هر یک از این چهار واکنش اختصاصی باز انتهای ۵’ مشترکی خواهند داشت که توسط پرایمر تعیین میشود. انتهای ۳’ در بین این قطعات متفاوت است، چون اتصال ddNTPها به صورت تصادفی و در یکی از نقاط فراوانی که قابلیت پذیرش آن باز را دارند انجام میشود.
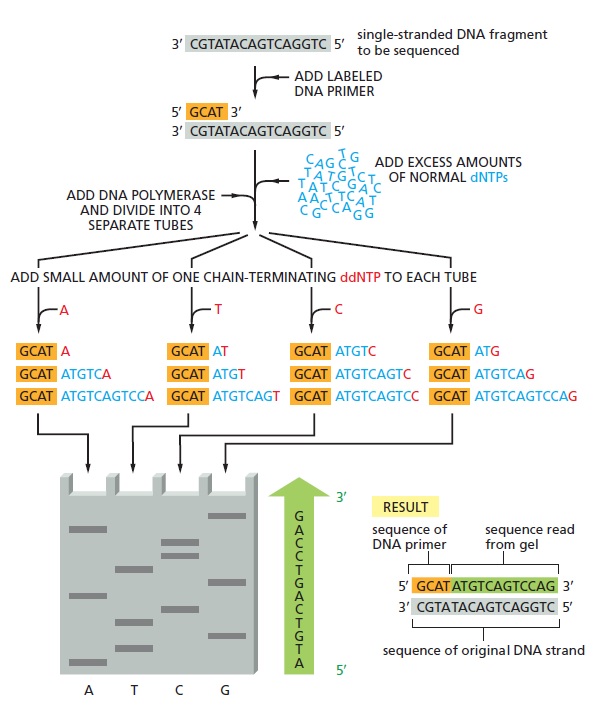 تصویر ۳ . به منظور تعیین توالی کامل این قطعه DNA تکرشتهای (خاکستری)، مولکول DNA ابتدا با یک پرایمر کوتاه (نارنجی) که با رنگ فلورسنت یا رادیوایزوتوپ لیبل شده است، هیبرید میشود. DNA پلیمراز و مقادیر فراوانی از هر ۴ نوع dNTP نرمال (آبی) به DNA متصلشده به پرایمر، افزوده میشوند و سپس مخلوط واکنش بین ۴ لوله جداگانه تقسیم میگردد. به هر کدام از این لولهها مقادیر اندکی از یک نوع ddNTP اضافه میشود (قرمز). چون در هر لوله مقادیر فراوانی از یک نوع توالی الگو وجود دارد و نوکلئوتیدهای خاتمه دهنده زنجیره تنها گاها به زنجیره در حال ساخت وارد میگردند، هر واکنش مجموعهای از نسخهها را داراست که در نقاط مختلفی از توالی خود خاتمه یافته اند. محصولات این ۴ واکنش توسط الکتروفورز و به صورت ۴ ستون موازی ژل پلی آکریل آمید از یکدیگر جدا میشوند. در هر ستون نوارها نشان دهنده قطعاتی هستند که در نقاط متفاوتی در نوکلئوتید موردنظر خاتمه یافته اند. با شروع از انتهای ژل و خواندن یک به یک تمام نوارها، توالی رشتههای سنتز شده DNA تعیین میگردد. توالی درج شده در فلش سبز مکمل توالی رشته اصلی (خاکستری) میباشد.
تصویر ۳ . به منظور تعیین توالی کامل این قطعه DNA تکرشتهای (خاکستری)، مولکول DNA ابتدا با یک پرایمر کوتاه (نارنجی) که با رنگ فلورسنت یا رادیوایزوتوپ لیبل شده است، هیبرید میشود. DNA پلیمراز و مقادیر فراوانی از هر ۴ نوع dNTP نرمال (آبی) به DNA متصلشده به پرایمر، افزوده میشوند و سپس مخلوط واکنش بین ۴ لوله جداگانه تقسیم میگردد. به هر کدام از این لولهها مقادیر اندکی از یک نوع ddNTP اضافه میشود (قرمز). چون در هر لوله مقادیر فراوانی از یک نوع توالی الگو وجود دارد و نوکلئوتیدهای خاتمه دهنده زنجیره تنها گاها به زنجیره در حال ساخت وارد میگردند، هر واکنش مجموعهای از نسخهها را داراست که در نقاط مختلفی از توالی خود خاتمه یافته اند. محصولات این ۴ واکنش توسط الکتروفورز و به صورت ۴ ستون موازی ژل پلی آکریل آمید از یکدیگر جدا میشوند. در هر ستون نوارها نشان دهنده قطعاتی هستند که در نقاط متفاوتی در نوکلئوتید موردنظر خاتمه یافته اند. با شروع از انتهای ژل و خواندن یک به یک تمام نوارها، توالی رشتههای سنتز شده DNA تعیین میگردد. توالی درج شده در فلش سبز مکمل توالی رشته اصلی (خاکستری) میباشد.
اتوماسیون تکنیک توالی یابی Sanger کارایی آن را افزایش میدهد
در سالهای ابتدایی انجام این تکنیک، دانشمندان مجبور بودند خوانش توالیها را به صورت دستی و از روی اتورادیوگرام انجام دهند. هر کدام از انواع باز در ستونی جداگانه قرار داشت و خوانش اتورادیوگرام از پایین به بالا انجام میشد. دادهها نیز به صورت دستی وارد کامپیوتر میشدند. همان طور که میتوانید تصور کنید، این پروسه بسیار طولانی بود. ۱۲ ساعت برای الکتروفورز، ۱۲ ساعت برای ایجاد اتورادیوگرام و مدت زمان بسیار طولانیتری برای خواندن توالیها موردنیاز بود. علاوه بر آن احتمال وقوع اشتباه نیز بالا و استفاده از بازهای لیبل شده با مواد رادیواکتیو خطرناک بود.
با توجه به موارد گفته شده، انجام تعدادی از اصلاحات ضروری بودند، خصوصا اگر هدف توالی یابی ژنومهای بزرگتری مانند ژنوم انسان بود. ماشینهای توالی یابی اتوماتیک که از رنگهای فلورسنت استفاده میکردند، در اوایل دهه ۱۹۹۰ به صورت تجاری موجود شدند. انجام فرایند توالی یابی به صورت ماشینی جلوگیری از وقوع اشتباهات را آسانتر میکرد و سریعتر و امنتر نیز بود. با اینکه به علت گران قیمت بودن دستگاه آنالیزکننده توالی، هزینه راهاندازی این تکنیک بسیار بالا است، اما از آن جایی که چندین نمونه به صورت همزمان آنالیز میشوند، هزینه توالی یابی هر کدام از نمونهها بسیار پایین است.
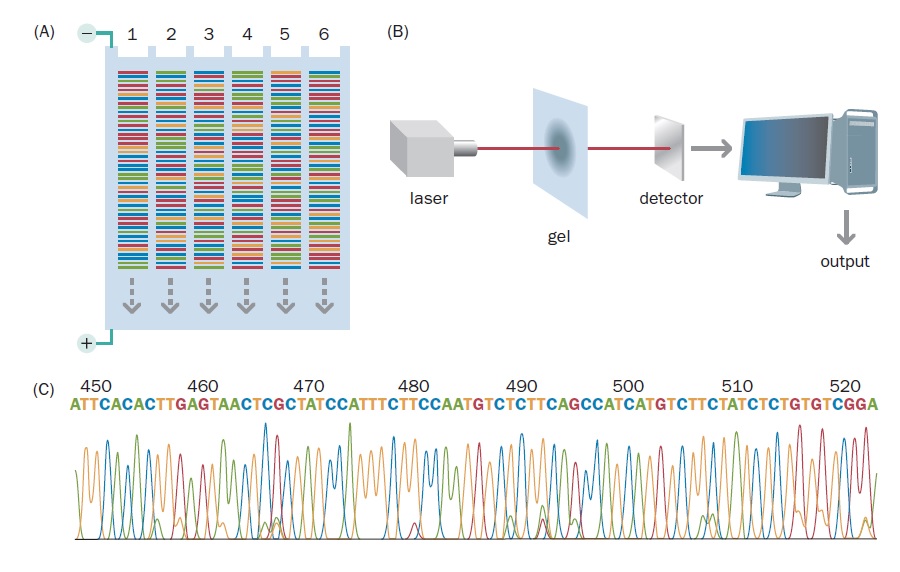
در این فرایند، مخلوط واکنش شامل ۴ نوع دئوکسی نوکلئوتید، ۴ نوع دی دئوکسی نوکلئوتید، یک پرایمر، DNA الگو و DNA پلیمراز میباشد. به منظور تمایز دی دئوکسی نوکلئوتیدها از یکدیگر، از چهار رنگ فلورسنت متفاوت برای هر یک از چهار واکنش اختصاصی باز استفاده میشود. با انتخاب رنگهایی که دارای طول موج نشری متفاوتی هستند، میتوان هر چهار واکنش را به صورت یک نمونه در چاهک ژل وارد کرد.
تکثیر DNA الگو توسط PCR و در دستگاه Thermal cycler صورت میگیرد. ابتدا DNA الگو دناتوره میشود. سپس دما پایین آورده میشود تا پرایمرها متصل شوند. در نهایت دما تا حد مناسب برای فعالیت DNA پلیمراز افزایش مییابد تا قطعات موردنظر تکثیر یابند. حین پلیمریزاسیون، ممکن است دی دئوکسی نوکلئوتیدها متصل شده و زنجیره DNA خاتمه پیدا کند. نسبت دی دئوکسی نوکلئوتیدها به دئوکسی نوکلئوتیدها به گونهای تنظیم میشود که توقف همانندسازی حتما در هر کدام از بازهای A، G، T و C صورت بگیرد.
پس از آن که پلیمراز از توالی الگو هزاران نسخه که هر کدام در نوکلئوتید متفاوتی خاتمه یافتهاند، تهیه کرد، کل مخلوط در یک ستون الکتروفورز میشود. هنگام الکتروفورز، قطعات DNA از منبع تحریکی مانند لیزر عبور میکنند. در این حین، همزمان با عبور قطعات DNA از نقطهای معین در ژل، سیگنالهای فلورسنت شناسایی و ضبط میگردند. نتیجه، ایجاد یک intensity profile برای هر کدام از فلوروفورها است که در آن، هر کدام از چهار رنگ نشان دهنده باز متفاوتی است. همزمان اطلاعات به صورت الکترونیکی ذخیرهسازی میشوند.
تکنیکهای اولیه از ژل slab پلی آکریل آمید استفاده میکردند، اما با استفاده از توالی یابی capillary دانشمندان توانستند ظرفیت فرایند توالی یابی DNA را به میزان زیادی افزایش دهند. در این تکنیک نمونههای DNA از طریق لولههای شیشهای موئین بلند و بسیار نازکی که قطرشان در حدود ۰.۱ میلیمتر است و حاوی ژل پلی آکریل آمید هستند، الکتروفورز میشوند. انجام این کار باعث حصول به درجات بالاتری از اتوماسیون میگردد. برخی از ماشینهای توالی یابی میتوانند توالی بیش از ۳۸۴ نمونه مختلف DNA را با استفاده از لولههای موئین پر شده از ژل تشخیص دهند.
تکنیک توالی یابی Sanger با تغییرات جزئی که در آن ایجاد شد، به مدت سه دهه ژنتیک مولکولی را پایهریزی و ژنوم انسان و بسیاری از ارگانیسمهای دیگر را توالییابی نمود. با این حال نقصهایی نیز داشت و از جمله مهمترین آنها وابستگی آن به الکتروفورز برای جداسازی قطعات جدید DNA بود. این کار نه تنها نیاز تکنیک به نیروی انسانی را افزایش میدهد، بلکه توالی یابی تعداد زیادی از قطعات DNA را به صورت همزمان دشوار میکند. درنتیجه کارایی توالی یابی محدود و تقریبا ۳۰ تا ۶۰ کیلوباز در هر ۳ تا۴ ساعت انجام الکتروفورز است. این تکنیک همچنین سرعت پایین و هزینه بالایی دارد.
 تصویر ۴ . ماشینهای کاملا اتوماتیک میتوانند واکنشهای توالی یابی دی دئوکسی را انجام دهند. در این تکنیکها dNTPهای نرمال در مقادیر بالا به همراه مخلوطی از چهار نوع ddNTP مورد استفاده قرار میگیرند. چهار رنگ فلورسنت جداگانه به عنوان لیبل در واکنشهای اختصاصی باز استفاده میشوند. لیبلها میتوانند به ddNTP اختصاصی باز اتصال یابند و یا اینکه به پرایمر متصل شوند و چهار نوع پرایمر متفاوت که چهار واکنش را از هم تمایز میدهند را داشته باشیم. نمونههای حاوی هر چهار واکنش توالی یابی توسط الکتروفورز در ژل پلی آکریل آمید بر اساس اندازه از همدیگر جدا میشوند. در این تصویر قطعات در ژل slab و در حال مهاجرت به سمت پایین نشان داده شدهاند. همزمان با مهاجرت قطعات به سمت پایین در حین الکتروفورز، پرتو لیزر بر روی نقطه ثابتی در ژل تابیده میشود و این کار باعث نشر رنگ فلورسنت از قطعات DNA در حال عبور از این نقطه میگردد. بیشترین فلورسنت هر کدام از رنگها در طول موجهای مختلفی رخ میدهد. اطلاعات به صورت الکترونیک ضبط و توالی در یک دیتابیس کامپیوتری ذخیره میگردد. هر قله رنگی نشانگر یک نوکلئوتید در توالی DNA است.
تصویر ۴ . ماشینهای کاملا اتوماتیک میتوانند واکنشهای توالی یابی دی دئوکسی را انجام دهند. در این تکنیکها dNTPهای نرمال در مقادیر بالا به همراه مخلوطی از چهار نوع ddNTP مورد استفاده قرار میگیرند. چهار رنگ فلورسنت جداگانه به عنوان لیبل در واکنشهای اختصاصی باز استفاده میشوند. لیبلها میتوانند به ddNTP اختصاصی باز اتصال یابند و یا اینکه به پرایمر متصل شوند و چهار نوع پرایمر متفاوت که چهار واکنش را از هم تمایز میدهند را داشته باشیم. نمونههای حاوی هر چهار واکنش توالی یابی توسط الکتروفورز در ژل پلی آکریل آمید بر اساس اندازه از همدیگر جدا میشوند. در این تصویر قطعات در ژل slab و در حال مهاجرت به سمت پایین نشان داده شدهاند. همزمان با مهاجرت قطعات به سمت پایین در حین الکتروفورز، پرتو لیزر بر روی نقطه ثابتی در ژل تابیده میشود و این کار باعث نشر رنگ فلورسنت از قطعات DNA در حال عبور از این نقطه میگردد. بیشترین فلورسنت هر کدام از رنگها در طول موجهای مختلفی رخ میدهد. اطلاعات به صورت الکترونیک ضبط و توالی در یک دیتابیس کامپیوتری ذخیره میگردد. هر قله رنگی نشانگر یک نوکلئوتید در توالی DNA است.



2 دیدگاه ها
[…] توالی یابی چیست؟ […]
[…] توالی یابی چیست؟ […]